 Uso: Clasificador probabilístico
Uso: Clasificador probabilístico
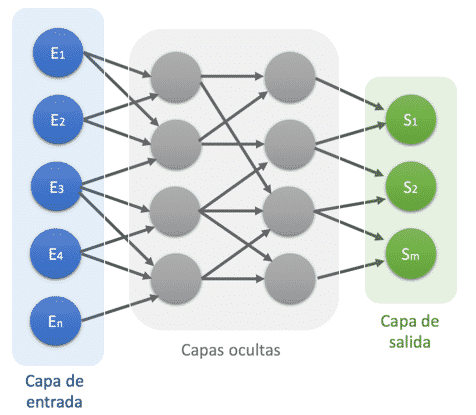
Descripción: Las redes neuronales son un paradigma de aprendizaje y procesamiento automático basado en el funcionamiento de nuestro sistema nervioso, este sistema este compuesto por una serie de neuronas y conexiones entre ellas que colaboran entre sí para producir una salida.
Variable dependiente: métricas y no métricas
Variables independientes: métricas y no métricas
Ejemplo en R: Clasificar tipo de flor atendiendo a sus características físicas como pueden ser el ancho y alto de los pétalos y sépalos.
# Carga de todos los paquetes necesarios#install.packages("ggplot2") #install.packages("lattice") #install.packages("caret") #install.packages("nnet") #install.packages("e1071") #install.packages("foreach") #install.packages("glmnet") library(ggplot2) library(lattice) library(caret) library(nnet) library(e1071) library(foreach) library(glmnet)# Carga de datos inicial, tipos de flores con diferentes características data(iris) View(iris) datos <- iris # Selección de una submuestra del 70% de los datos para entrenamiento y el 30% se usará para testeo del método set.seed(101) tamano.total <- nrow(datos) tamano.train <- round(tamano.total*0.7) datos.indices <- sample(1:tamano.total , size=tamano.train) datos.train <- datos[datos.indices,] datos.test <- datos[-datos.indices,] # Previamente se usará train para ver el valor de los parámetros size y decay # size: número de unidades ocultas intermedias # decay: se usa para evitar el sobre ajuste parametros <- train(Species~., data=datos.train, method="nnet", trace=F) size <- parametros$bestTune$size decay <- parametros$bestTune$decay parametros$bestTune
## size decay
## 6 3 0.1# Entrenamiento de la red neuronal con los valores de train
modelo <- nnet(Species ~ ., size=size, decay=decay, trace=F, data=datos.train)
modelo## a 4-3-3 network with 27 weights
## inputs: Sepal.Length Sepal.Width Petal.Length Petal.Width
## output(s): Species
## options were - softmax modelling decay=0.1# Predicción. Se crea un data frame con las probabilidades y los nombres de las especies
predicciones <- data.frame(predict(modelo, datos.test), Specie=predict(modelo,datos.test, type="class"))
predicciones## setosa versicolor virginica Specie
## 5 0.984696025 0.01467717 0.0006268079 setosa
## 10 0.979163054 0.02002491 0.0008120312 setosa
## 12 0.979718900 0.01948811 0.0007929853 setosa
## 15 0.987746650 0.01173706 0.0005162860 setosa
## 16 0.987281279 0.01218561 0.0005331137 setosa
## 18 0.983802343 0.01553962 0.0006580417 setosa
## 22 0.983603181 0.01573255 0.0006642712 setosa
## 23 0.986727467 0.01271700 0.0005555347 setosa
...# Matriz de confusión
mc <- table(predicciones$Specie,datos.test$Species, dnn = c("Asignado","Real"))
mc## Real
## Asignado setosa versicolor virginica
## setosa 15 0 0
## versicolor 0 12 0
## virginica 0 0 18# Porcentaje de aciertos
aciertos <- sum(diag(mc)) / nrow(datos.test) * 100
aciertos## [1] 100
Nota: Consular la implementación de una red neuronal convolucional en R con keras




Gracias!!! muy buena explicación desde hoy seré segudidor de tu web,
saludos desde Bogotá ,Colombia
Estoy encantado de que te sea de utilidad.
Seguiré publicando articulos sobre este tema, puesto que es el tema central de mi tesis doctoral.
Un saludo desde España!!!
Excelente explicación, ,e gusta mucho tu pagina y tu trabajo.
Podrias subir algun script en R de redes neuronales recurrentes LSTM que tenga algunas variables categoricas, seria de gran ayuda. Gracias, saludos desde Colombia.
En este momento tengo el foco puesto en la parte de Big Data, cuando vuelva sobre este tema tendré en cuenta tu comentario. De todas maneras si algún lector tiene un ejemplo no dude en contactar en info@diegocalvo.es y le publicaré el articulo. Por supuesto bajo su autoría y como colaborador.
Excelente.
Buenas estimado, muchas gracias por tu tutorial, pero me gustaria que me puedas guiar en algo, estaria muy agradecido, es sobre como puedo analizar imagenes de trayectorias vehiculares usando redes neuronales. Si puedes responderme para poder hablar mejor del tema.
Saludos